【9.2.1】IVT(mRNA合成|In Vitro Transcription)
RNA合成可以分为固相化学合成和酶法合成。
一、Solid Phase Chemical Synthesis
固相化学合成适用于一些短链RNA合成(50-100nt)。不足的地方在于,随着RNA的长度增加,合成产量和纯度会发生下降。申基生物研发人员称,他们可以采用固相化学合成mRNA的长度达到150bp,如果去掉20bp的polyA尾巴,此150bp mRNA可编码产生40个氨基酸的多肽序列。此类短肽的mRNA序列可以用于表达抗原表位肽,进行疫苗研发。
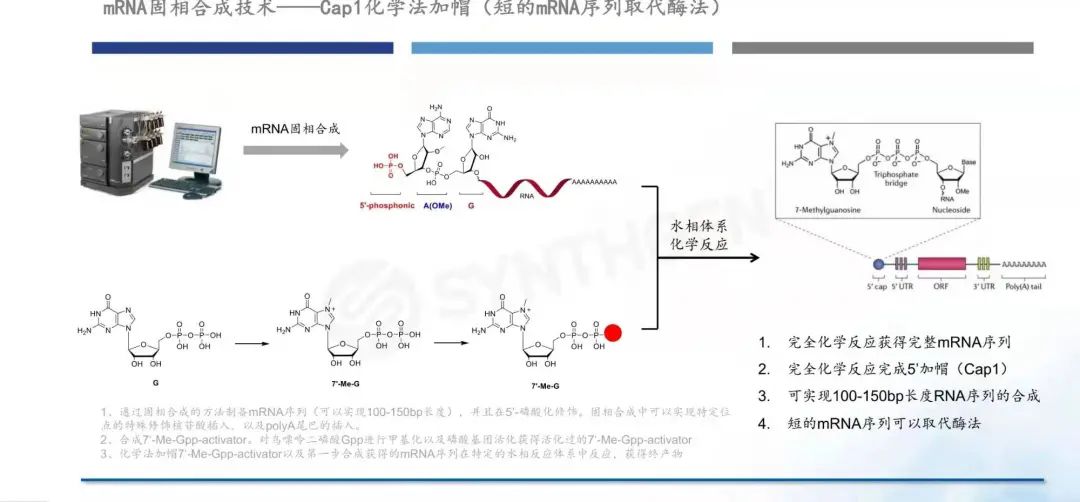
mRNA固相合成方法
二、Enzymatic Synthesis
适用于各种长度RNA,实现高产量,还得靠酶学的方法。酶法合成利用T7 RNA polymerase ,通过体外转录反应来实现高产量的各种长度的RNA合成。T7 RNApolymerase 在体外也具有有启动子特异性的转录活性,是第一个被鉴定出来的单亚基RNA polymerase,可在大肠杆菌种过表达,用于构建体外转录系统。早在八十年代左右,人们就把外源目的基因连到质粒上,提取质粒,酶切得到线性化的模板,以此线性化的质粒模板来合成RNA。
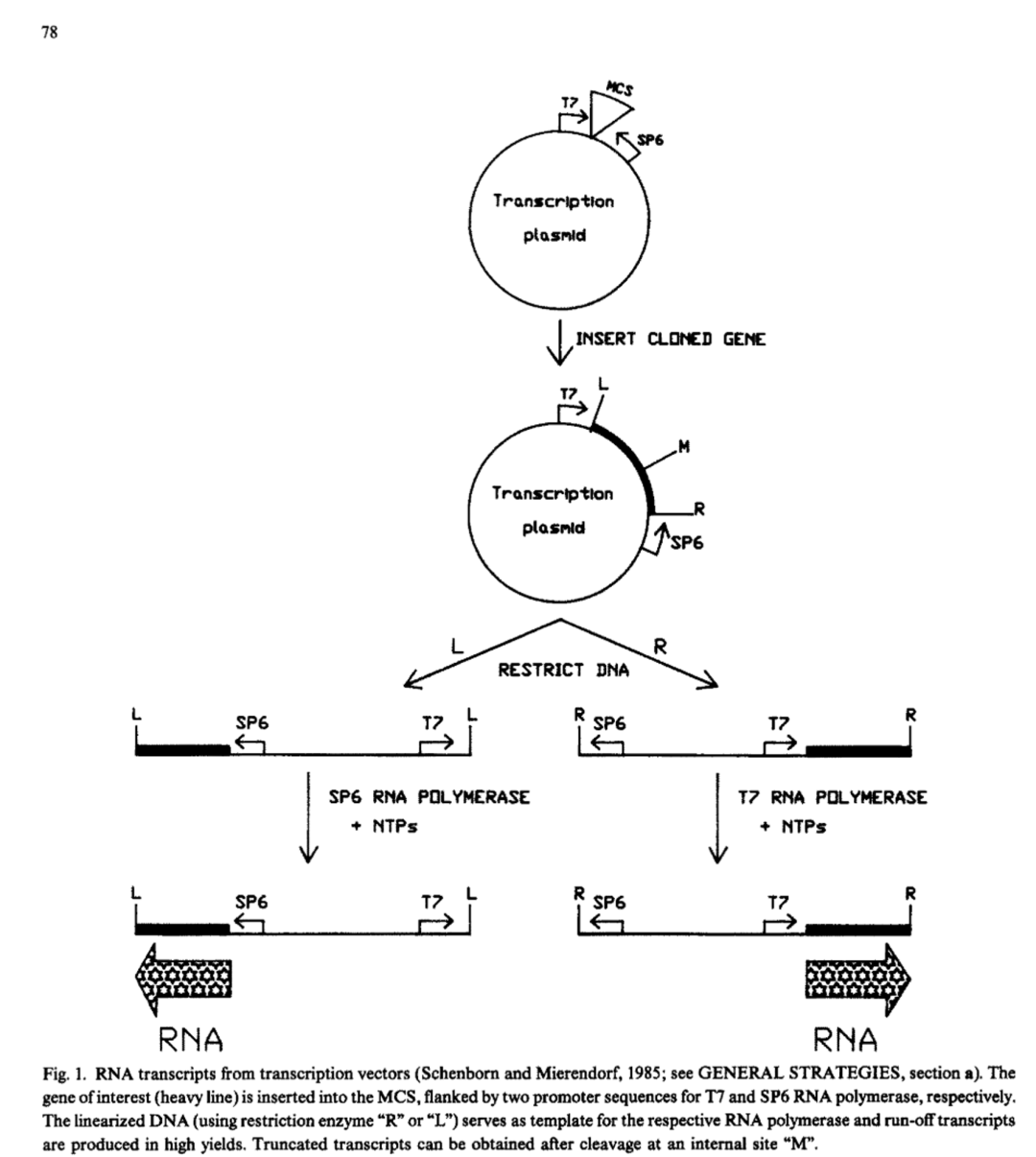
mRNA酶法合成
2.1 转录的基本原理
在转录起始位点上游,有一段保守启动子序列。RNA polymerase 识别并且结合到特异性的启动子上,打开转录起始位点附近的DNA双链,启动RNA的从头合成。RNA polymerase 利用NTPs起始转录,初始阶段,转录是不稳定低效的,典型特征是重复发生流产性起始事件,形成长度2-6 nt流产短链RNAs。一旦合成出超过10-12 nt RNA,RNA polymerase 构象发生改变,逃离启动子,DNA-酶-RNA形成稳定的三元复合物,进入高效转录阶段,直到RNA polymerase 遇到终止子或者从线性DNA模板的末端滑落掉下,mRNA转录合成便终止。
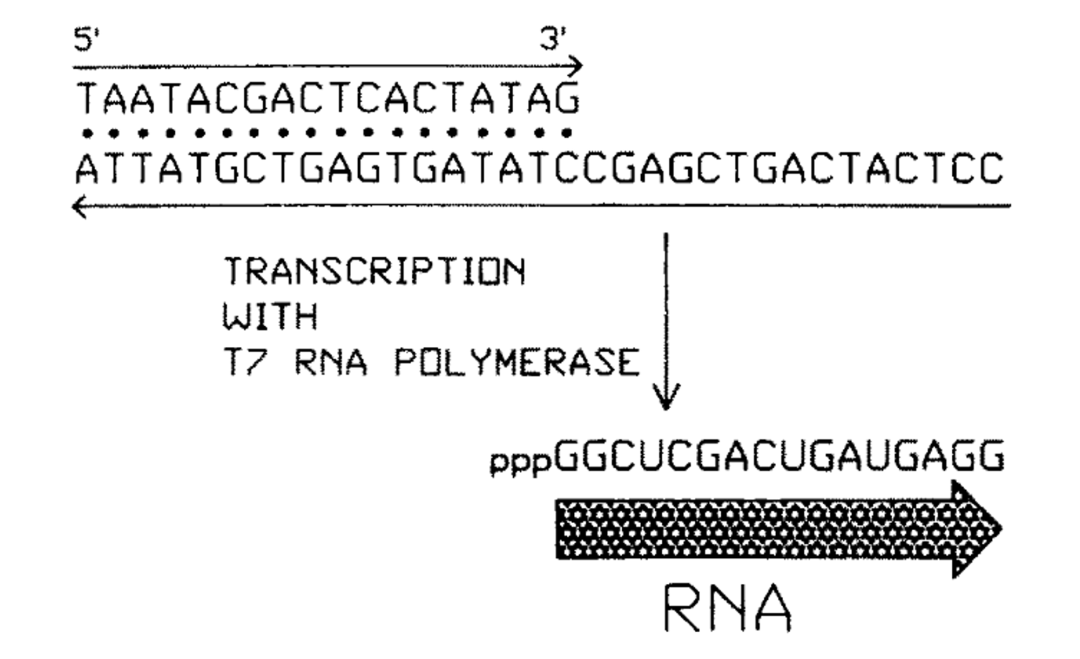
体外转录基本原理
2.2 T7 RNA polymerase Structure
细胞和叶绿体的 RNA polymerase 由多个不同的亚基组成,不同的亚基执行不同的功能。许多噬菌体和线粒体的RNA polymerase由单一的亚基构成,同时拥有多种功能,比如RNA的聚合,DNA双链的解开,DNA-RNA链的解开。
T7 RNAP主要32个 ɑ螺旋和9个 β折叠片构成。C端结构域有2/3跟Klenow fragment相似,由finger,thumb,palm,组成,N端结构域(1-325)是T7 RNA polymerase特有的,涉及启动子识别和DNA解链。T7 RNA polymerase可以解开-1 to -4 的碱基对,该区域的模板链置于含有催化活性位点的裂缝区域,形成一个transcription bubble。在promoter DNA双链邻近的大小沟,T7 RNA polymerase来实现启动子双链区域的序列特异性识别。Intercalating β hairpin(230-240)插入到DNA大沟里,实现碱基的直接识别。由740-770残基形成specificity loop同样可以识别碱基对。N端结构域通过插入一段弹性的loop结构(93-101)来识别DNA小沟中富含AT碱基的DNA序列(-17——13)。

体外转录基本原理
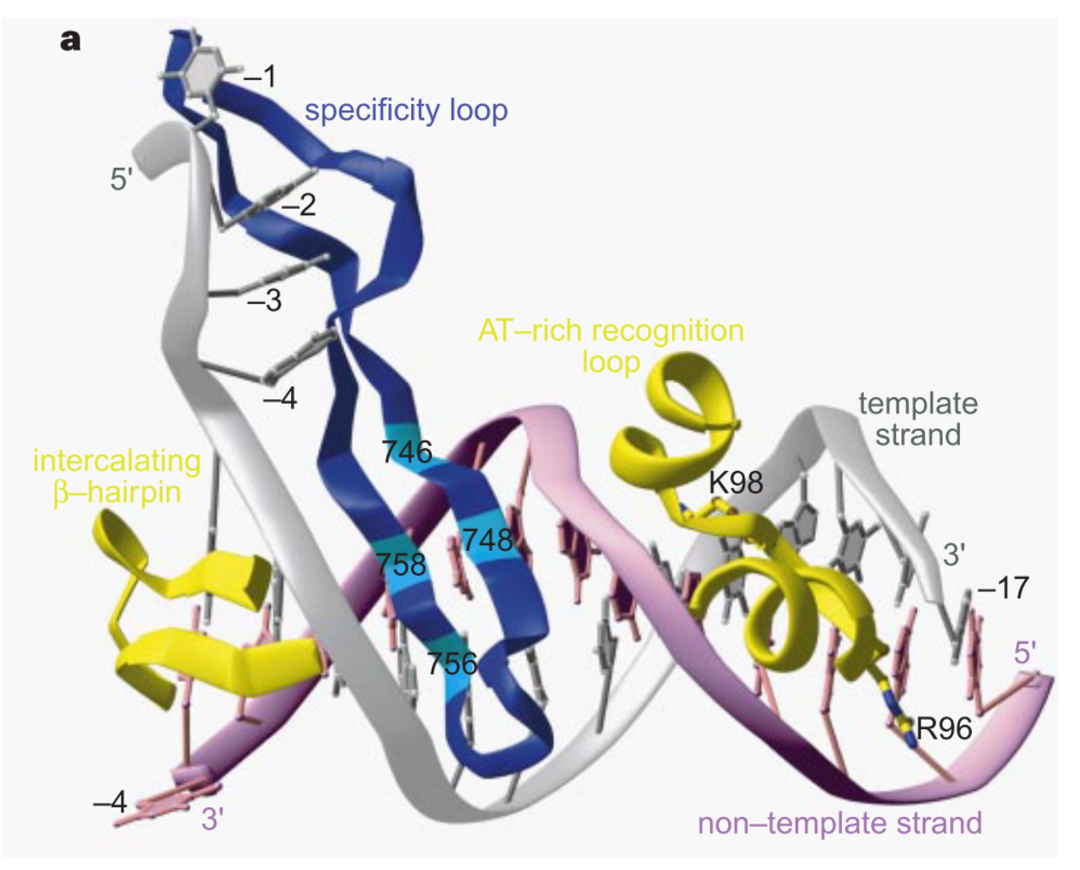
T7RNP和启动子序列之间的相互作用
转录初始和延伸阶段,在构象上,T7 RNA polymerase有由非常显著的差异。DNA-T7 RNA polymerase在延伸阶段非常稳定。T7 lysozyme可以抑制T7 RNA polymerase从初始阶段向延伸阶段的转变,但是对于转录延伸阶段的T7 RNA polymerase活性没有任何的影响。从转录初始进入到延伸阶段以后,T7 RNAP的N端结构域发生重排,破坏掉启动子结合结构域,创造出一个可以容纳7bp DNA-RNA异质性双链的通道,在异质性双链脱落以后,允许RNA转录本通过。
2.3 依赖启动子的聚合酶活性
T7 RNAploymerase同时有依赖于DNA模板和RNA模板的RNA聚合酶活性,可以高度特异性地识别保守启动子序列,但其聚合酶活性具有非常大的弹性尺度范围。不管双链DNA启动子序列后面跟着的是双链or 单链DNA序列,双链or单链RNA序列,chemeric DNA-RNA,T7 RNAploymerase 都可以启动转录合成RNA序列,这可以看作是T7 RNApolymerase 依赖于启动子的聚合酶活性。说白了,只要存在启动子DAN双链序列,不管后面接的是什么,T7 RNAploymerase都可以利用NTPs合成出产物RNA。
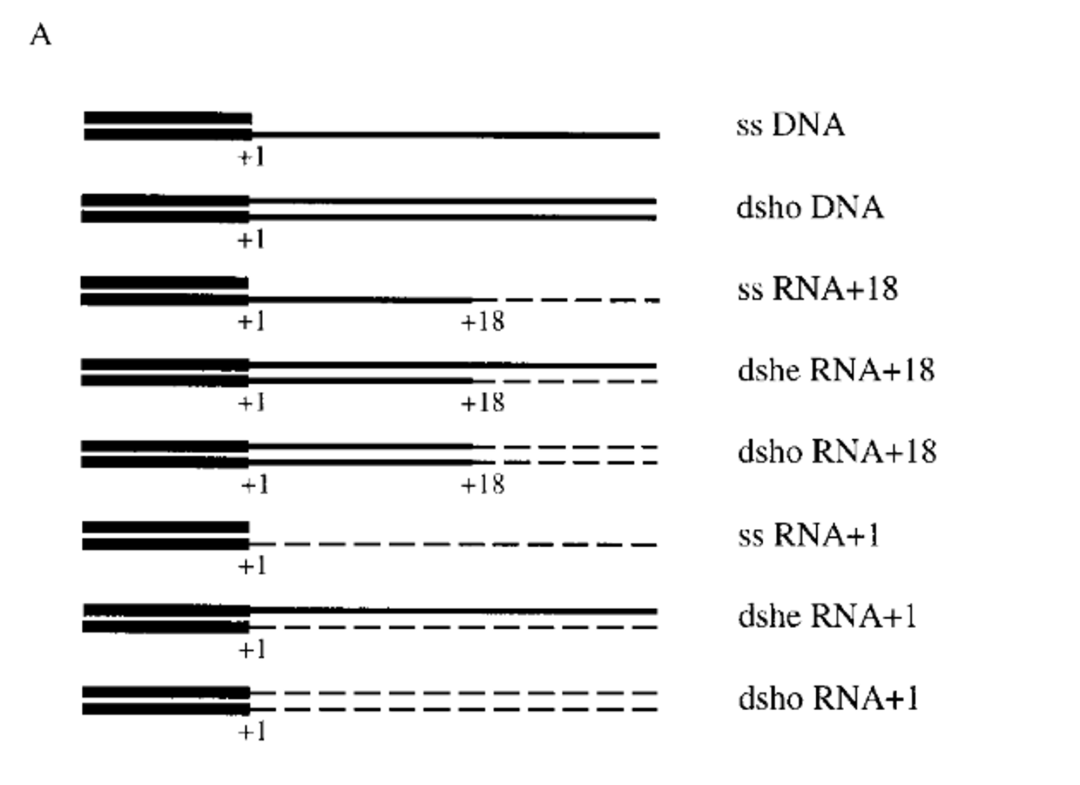
拥有启动子双链DNA序列的各种不同模板
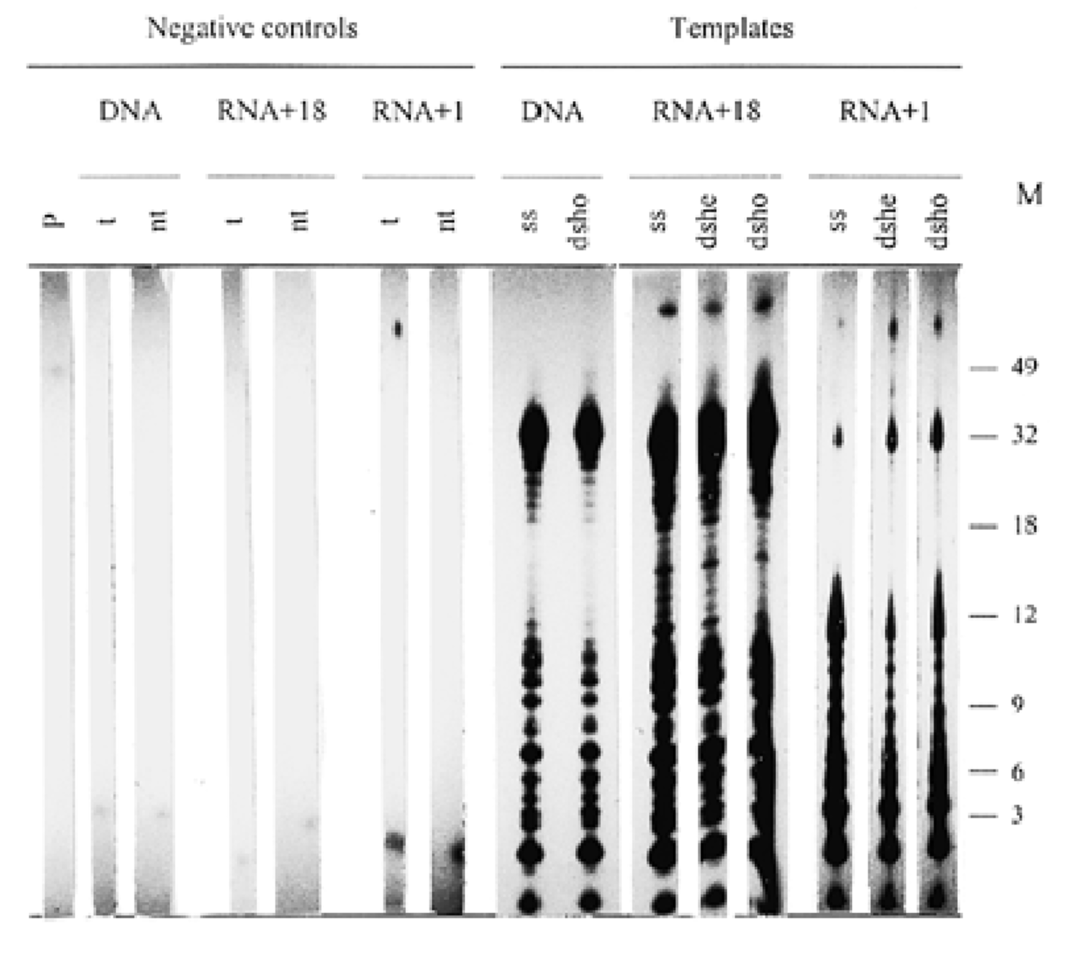
有启动子双链DNA序列的各种不同模板均可合成RNA
2.4 不依赖启动子的聚合酶活性
有研究利用RNA -Seq来研究体外转录反应产物序列和产量分布情况,发现IVT反应中的RNA产物末端可以折回形成发卡结构,将自身变成引物,在3’端添加NTPs,产生长度超过预期产物的RNA,并且随着RNA产物浓度的升高,此过程会跟以DNA为模板的正常转录过程发生竞争。以上过程,可以看作是独立于启动子序列的聚合酶活性。说白了,就是IVT反应的产物RNA可能会自己折叠形成发卡结构,把自己变成RNA引物,并且以自己为模板在末端添加核苷酸,这样的话就产生了很多超过预期长度的产物RNA。这个过程可能会循环发生,导致更长的RNA产物反而成了IVT反应的主要产物。
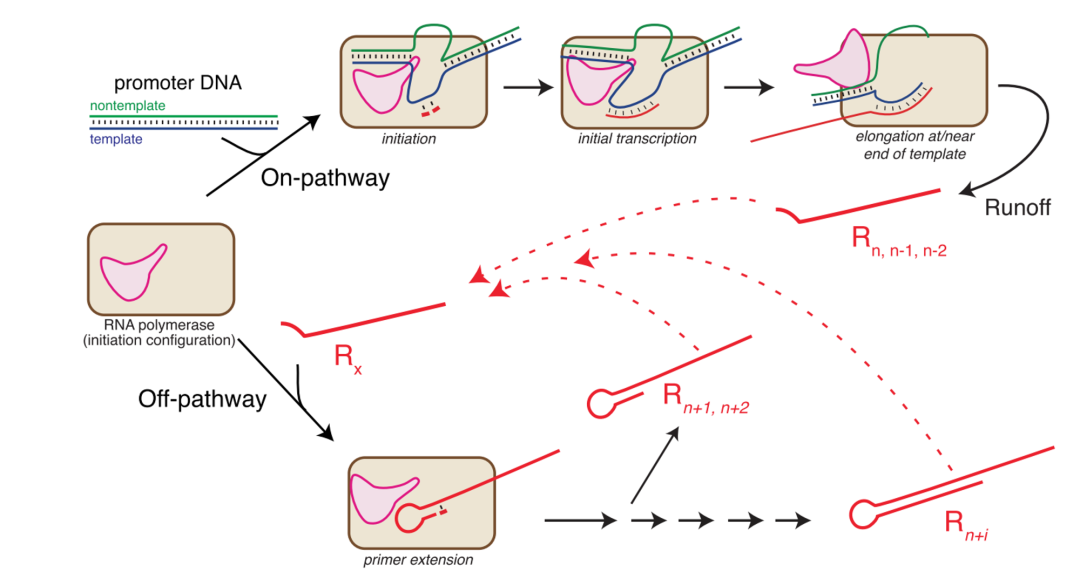
IVT反应中超过预期长度的mRNA形成机制
2.5 副产物给mRNA纯化增添困难
T7 RNA polymerase 在体外转录反应中,不仅可以产生符合预期长度的mRNA,还会产生一些副产物RNA,比如短的流产RNA(n-i),甚至更长的RNA(n+i)。IVT反应中,这些副产物增加了纯化目标mRNA的难度。IVT反应副产物RNA可能形成的dsRNA,会导致机体产生强烈的免疫反应,因此必须去除dsRNA。目前mRNA主流纯化工艺都是采用oligo dT柱子(只选择性地结合有polyA尾巴RNA),高盐浓度下,可以抑制携带负电荷的柱子配体和RNA之间的相互排斥,促进氢键形成。将盐离子去除以后,恢复柱子配体-RNA之间的负电荷之间的排斥,摧毁氢键,把RNA洗脱下来。
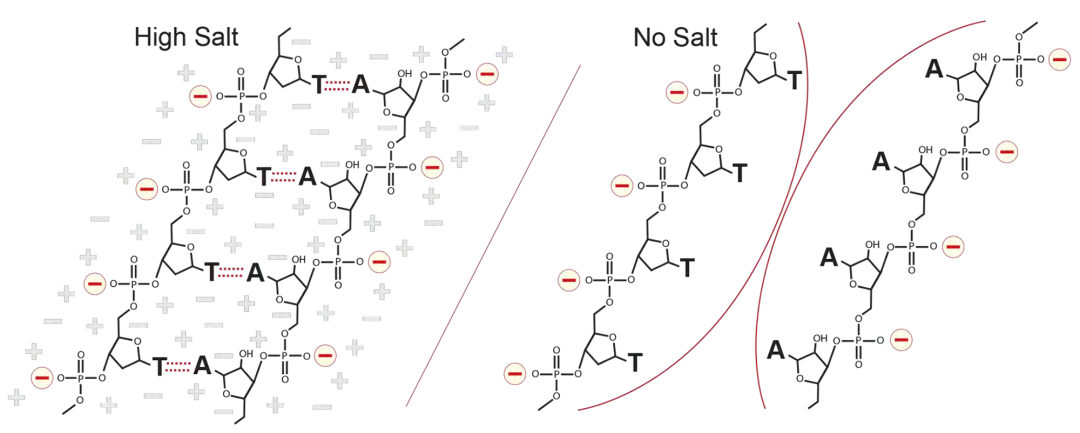
oligo dT柱子
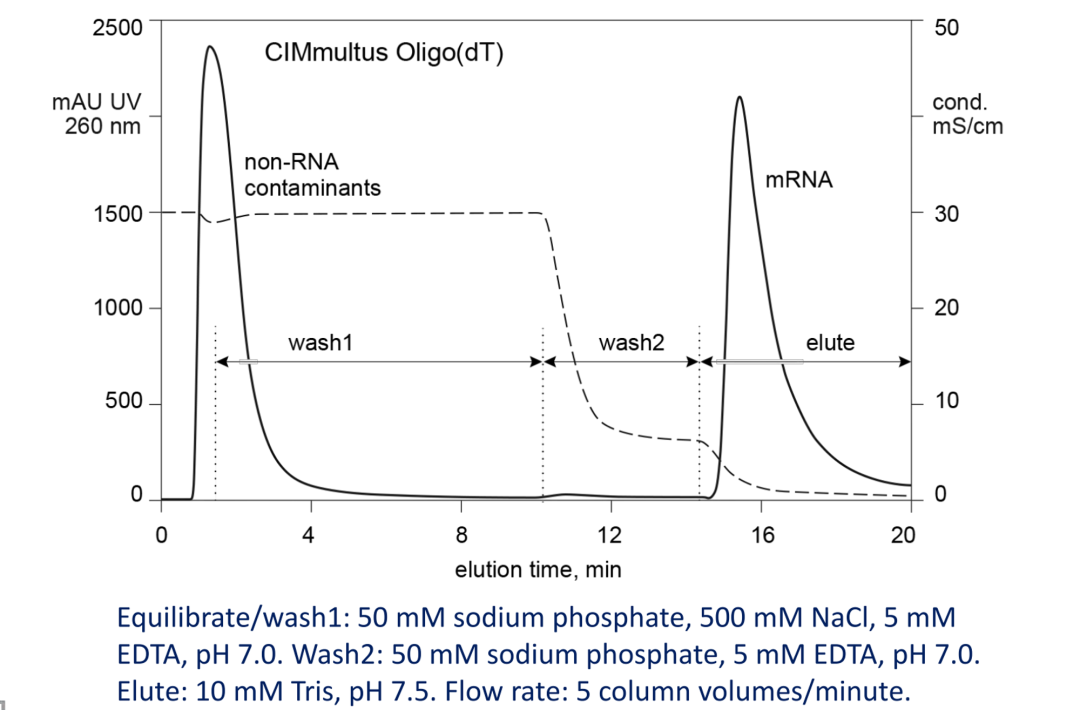
BIA柱子纯化mRNA出峰图
参考资料
