【4.2.3】LinearFold
网页工具: http://rna.baidu.com/
一、
二、算法
本质上,我们使用动态规划算法来将搜索空间从指数减小为多项式。我们首先使用确定性有限自动机(DFA)(一种带有标记的边缘以及不同的起始和结束节点)的有向图来表达氨基酸和蛋白质。下图显示了氨基酸DFA表示形式的四个示例,每个代表一个氨基酸
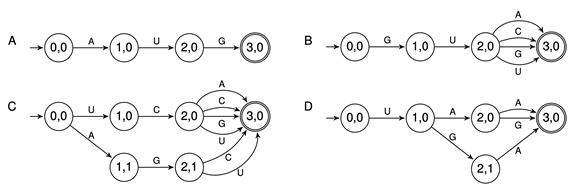
接下来,我们将它们连接到一个蛋白质序列p的单个DFA D(p)中,该序列表示翻译成该蛋白质D(p)= D(p1)◦D(p2)◦···D(pm)◦D(STOP)的所有可能的mRNA序列,方法是将每个DFA的末端节点与下一个DFA的起始节点缝合在一起。
我们需要通过DFA找到具有最稳定二级结构的mRNA序列。在这里,我们从计算语言学,随机上下文无关文法(SCFG,stochastic context-free grammar)中借用了一种工具,该工具可用于表示RNA折叠。现在,mRNA设计问题是单序列折叠问题在多输入情况下的简单扩展。我们在所有可能的序列的所有可能的结构中找到最小的自由能结构(及其对应的序列)。这可以通过在蛋白质DFA上交叉SCFG来解决。
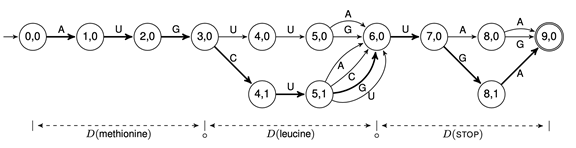
实际上,mRNA疫苗序列设计的优化是将单个RNA序列的二级结构计算(RNA折叠)扩展到多个RNA序列。在用DFA提取多个RNA序列后,我们通过采用DFA和SCFG的交集,从多个mRNA序列中找到具有最稳定二级结构的序列。
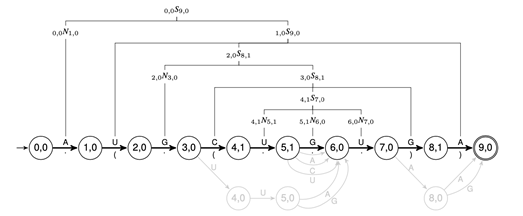
下图显示了DFA和SCFG如何相交以生成“蛋氨酸亮氨酸终止”序列为“ AUGCUGUGA”的示例。
在此基础上,我们的算法也从以下几个方面进行了扩展:
- 借用LinearFold的思想,进一步将计算复杂度从三次复杂度降低到线性度,从而大大减少了设计mRNA序列所需的时间;
- 从提供最佳的mRNA序列到提供前k个次优的mRNA序列作为选择。疫苗公司可以从这些替代方案中选择最合适的疫苗序列;
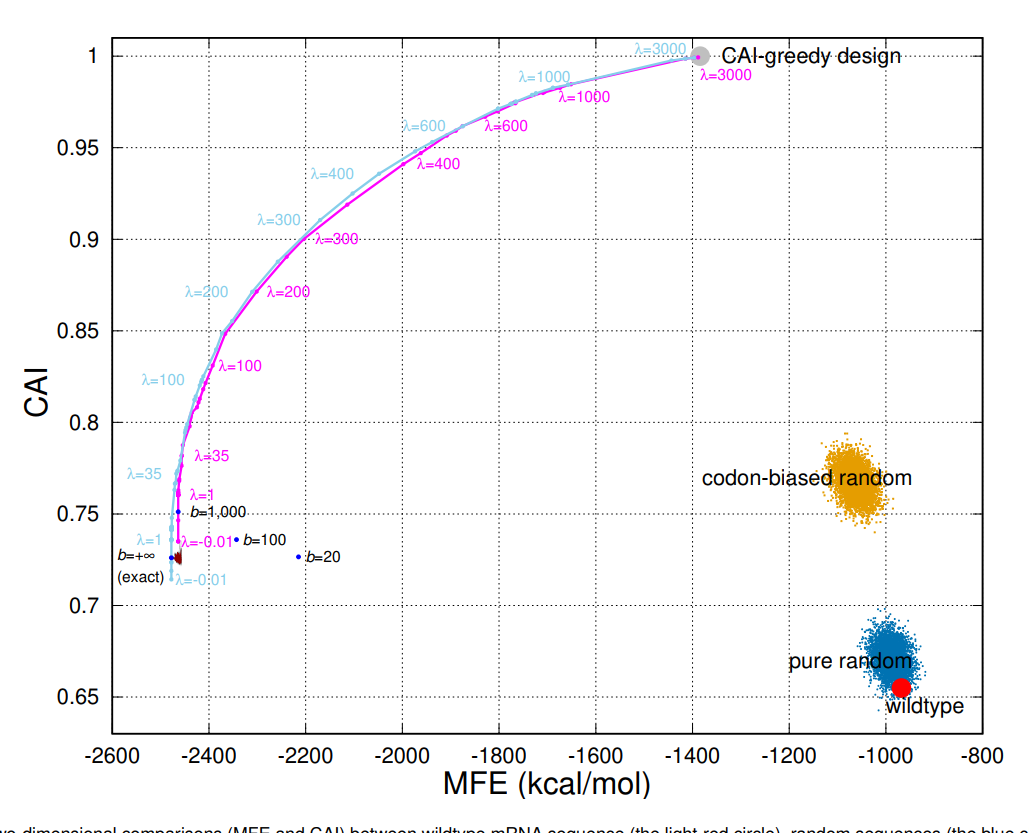
- 同时优化二级结构的稳定性和密码子的优化性,设计出稳定性好,蛋白表达效率高的mRNA疫苗序列。

参考资料
这里是一个广告位,,感兴趣的都可以发邮件聊聊:tiehan@sina.cn
![]() 个人公众号,比较懒,很少更新,可以在上面提问题,如果回复不及时,可发邮件给我: tiehan@sina.cn
个人公众号,比较懒,很少更新,可以在上面提问题,如果回复不及时,可发邮件给我: tiehan@sina.cn

