【9.1.2】moe叠合两分子结构
一、目标:叠合两分子结构

二、注意
- 在moe中输入时候,不要是中文输入法
三、流程
1.打开pdb文件
- File – open—pdb –输入pdb id
- File-open – pdb文件
2.序列注释
点击SEQ,
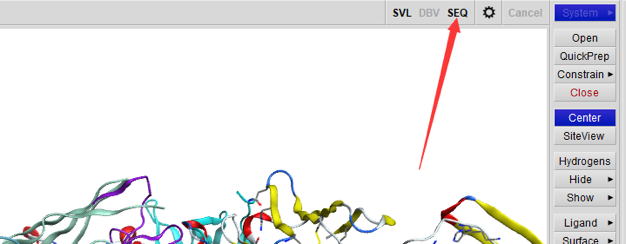
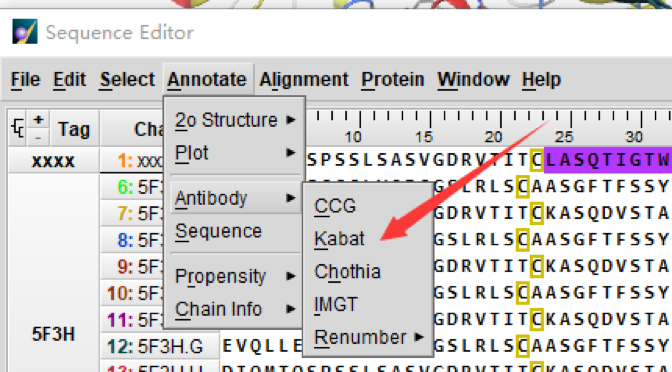
出现新的操作界面(如下图) 然后选择‘annotate’—‘antibody—‘kabat’
经过抗体,抗原注释,出现的结果是这样的
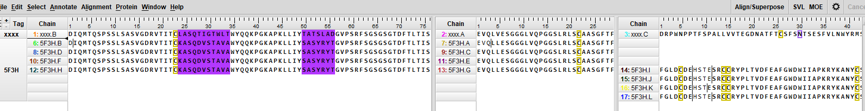
第一部分是重链,第二部分是轻链,第三部分是抗原区域
3. 序列叠合
然后点击界面右上角的“Align/Superpose”, 界面右下角出现各种选择框,点击红色箭头所示的箭头

用control加鼠标选中所需的行,被选中的行的序列名字背景色是灰色(如下图);然后通过setblocks 中I /* 来标注,* 表示被选中;i表示丢弃。然后点击”superpose”,被选中的序列则进行重拍
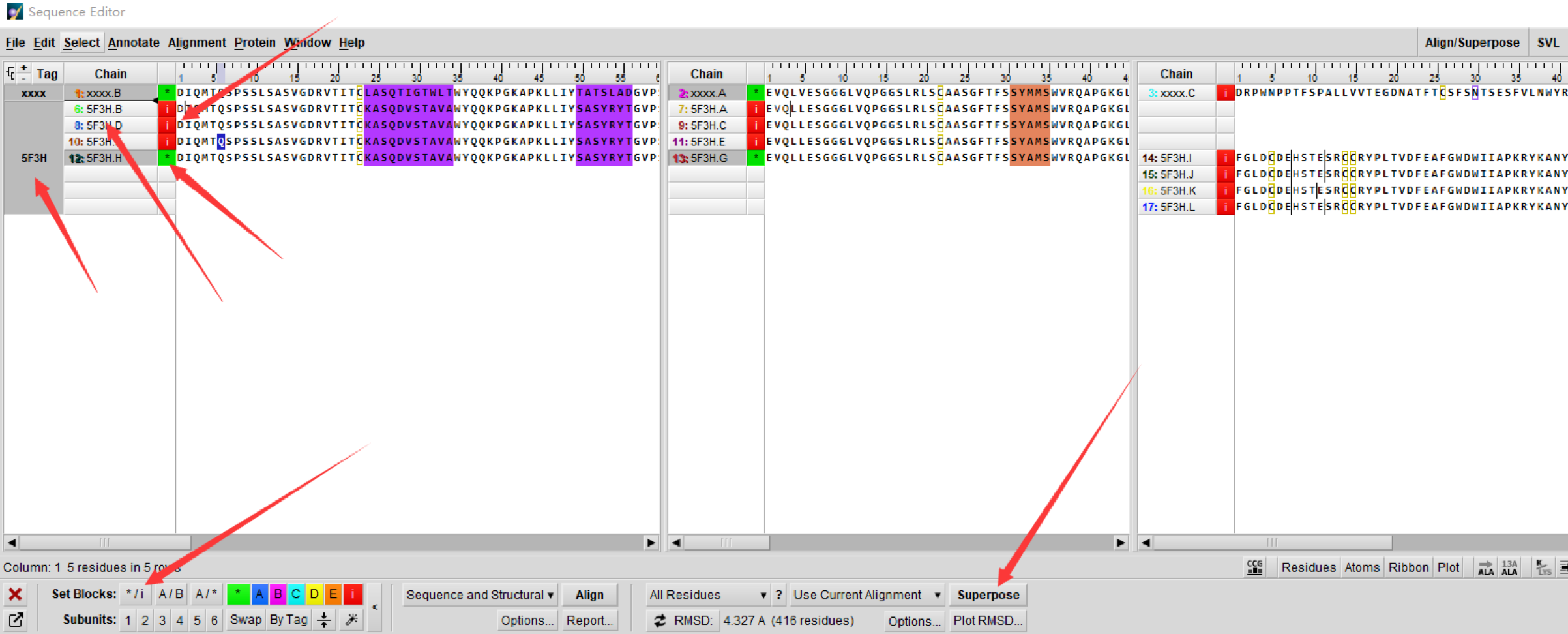
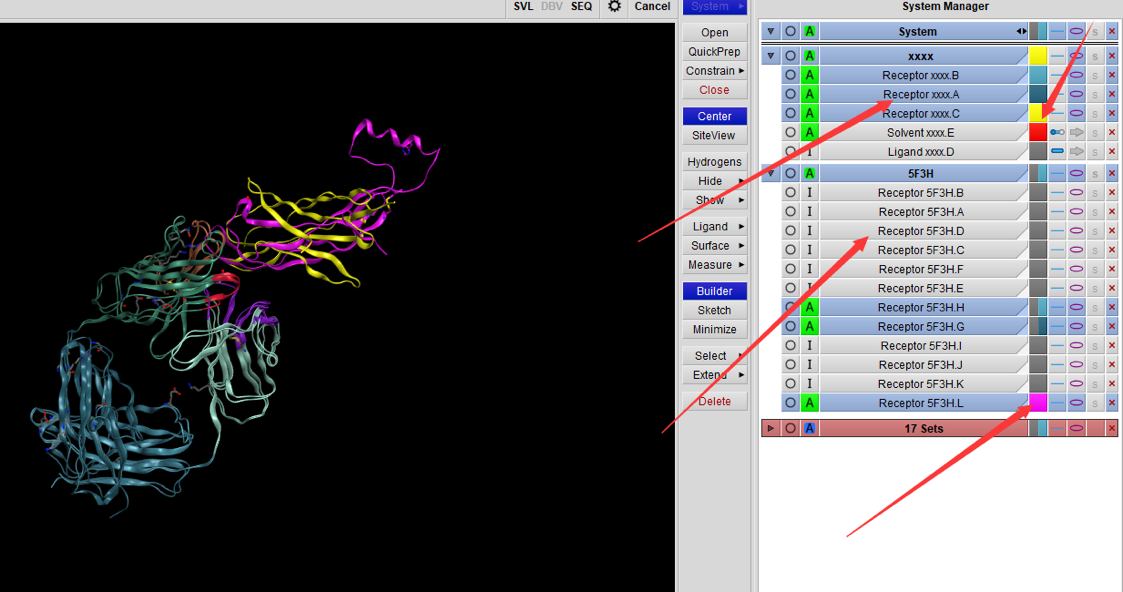
4.选择需要显示的序列
选中显示的链,并标注颜色。(如下图)背景色是绿色的部分,表示被选中,灰色的背景表示没有被选中(则结构不显示在图中),然后选择背景色。
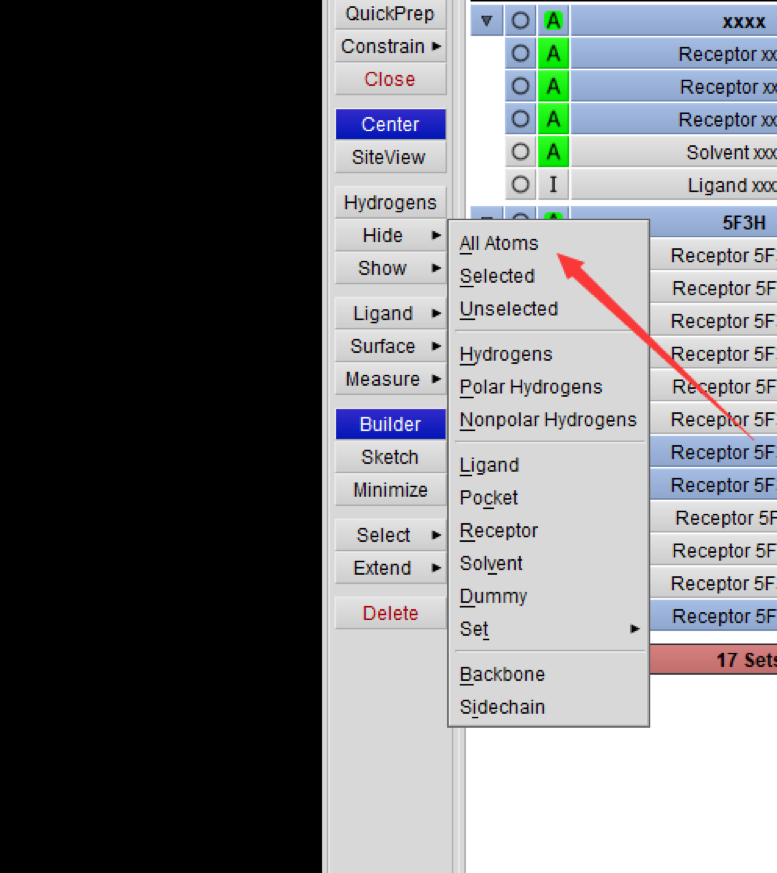
5.隐藏atoms
“Hide”—“All Atoms
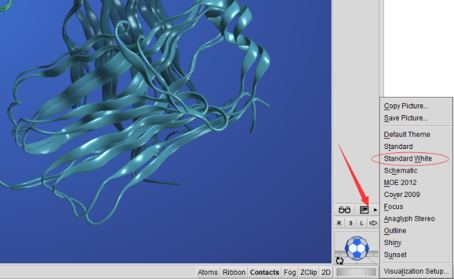
6. 背景选择白色
选择右下角的“相框”图标,然后选择背景“standard white”
7.保存结果
File – save – 输入保存的名字,保存为moe文件,以后就可以调取出来使用
这里是一个广告位,,感兴趣的都可以发邮件聊聊:tiehan@sina.cn
![]() 个人公众号,比较懒,很少更新,可以在上面提问题,如果回复不及时,可发邮件给我: tiehan@sina.cn
个人公众号,比较懒,很少更新,可以在上面提问题,如果回复不及时,可发邮件给我: tiehan@sina.cn

