【3.3.2】tablet可视化比对结果
软件官网:https://ics.hutton.ac.uk/tablet/
一、tablet安装
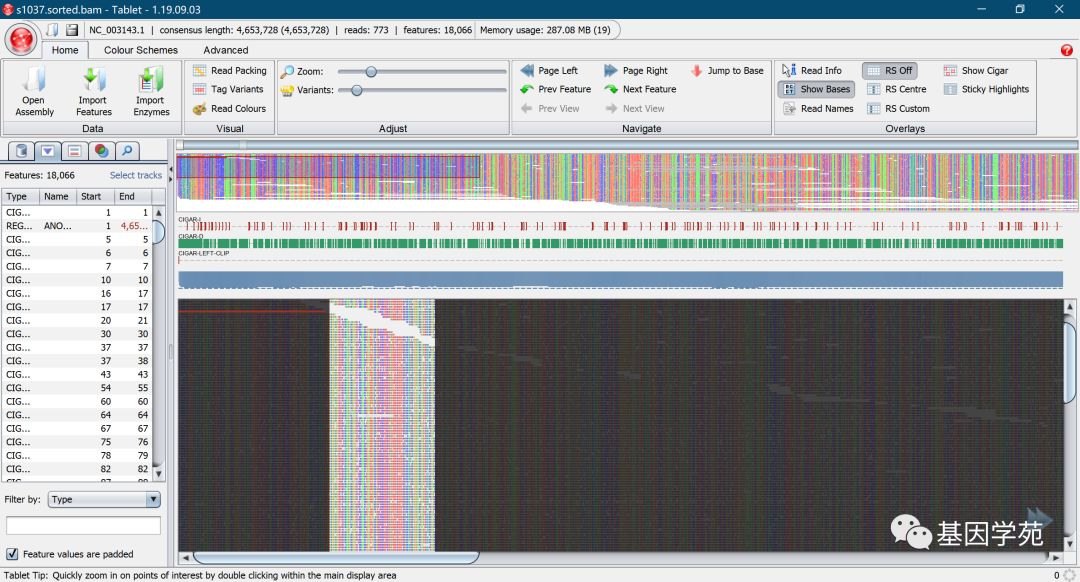
tablet可以用于基因组的可视化,例如可以查看拼接结果每个位点的细节信息,例如有多少位点覆盖以及每个位点具体的细节信息,还包括一些插入缺失的信息,通过tablet的可视化,可以用来评估拼接结果的准确性。tablet需要输入拼接基因组序列与测序数据与拼接结果比对的bam文件,输入的bam必须是排序建索引后的bam,同时拼接的基因组序列也需要建立索引,tablet工具支持windows,Linux,macos等多个版本。

图 1 tablet可视化基因组
二、使用
2.1 准备输入文件
#建立索引
minimap2 -d co92.min co92.fa
#比对
minimap2 -ax map-ont co92.min ../4.filter/clean.filtlong.fq.gz >s1037.sam
#samtools处理
samtools sort -@ 4 -O bam -o s0137.sorted.bam s1037.sam
samtools index s0137.sorted.bam
samtools faidx s1037.fa
2.2 使用案例
1、将准备好的文件,至少四个,参考序列fasta格式及索引fai文件,比对并排序后的bam文件及索引bai文件;
co92.fna
co92.fai
co92.gff
s1037.sorted.bam
s1037.sorted.bam.bai
2、打开tablet,首先导入bam文件,然后导入fasta文件,索引文件无需导入,但必须与对应文件在同一目录下,(tablet对中文支持不好);

3、可以通过鼠标调整显示模式;

4、tablet还支持导入gff格式结果,方便查看固定区域,(图中高亮区域)
参考资料
这里是一个广告位,,感兴趣的都可以发邮件聊聊:tiehan@sina.cn
![]() 个人公众号,比较懒,很少更新,可以在上面提问题,如果回复不及时,可发邮件给我: tiehan@sina.cn
个人公众号,比较懒,很少更新,可以在上面提问题,如果回复不及时,可发邮件给我: tiehan@sina.cn

