【1.1】打分矩阵
一、核酸序列的替换记分矩阵
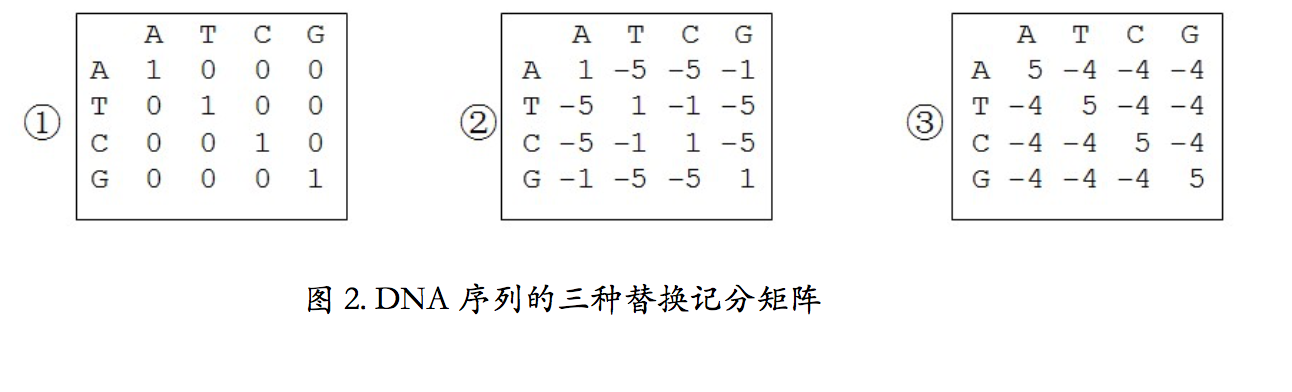
DNA 序列的替换记分矩阵主要有三种。一个是等价矩阵(图 1-1)。这个矩阵最简单. 其中,相同核苷酸之间的匹配得分为 1,不同核苷酸间的替换得分为 0。由于不含有碱基的 理化信息和不区别对待不同的替换,在实际的序列比较中很少使用,一般只用于理论计算。
另一种是转换-颠换矩阵(图 1-2)。这里就需要先复习下转换和颠换的概念了。我们说 核酸的碱基按照环结构特征被划分为两类,一类是嘌呤,包括腺嘌呤 A 和鸟嘌呤 G,它们 都有两个环;另一类是嘧啶,包括胞嘧啶 C 和胸腺嘧啶 T,它们只有一个环。如果 DNA 碱 基的替换保持环数不变,则称为转换,比如腺嘌呤 A 替换为 鸟嘌呤 G、或者胞嘧啶 C 替换 为胸腺嘧啶 T,也就是嘌呤变嘌呤,嘧啶变嘧啶;如果环数发生变化,则称为颠换,比如腺 嘌呤 A 替换为胞嘧啶 C、或者胸腺嘧啶 T 替换为鸟嘌呤 G,也就是嘌呤变嘧啶,或者嘧啶变嘌呤。在进化过程中,转换发生的频率远比颠换高。也就是说,大自然更倾向于接受嘌呤 和嘌呤之间的替换,以及嘧啶和嘧啶之间的替换,而嘌呤和嘧啶之间的替换会导致不好的事 情发生,这种替换大多在进化过程中已经被淘汰了。为了反映这一情况,转换-颠换矩阵中, 转换的得分比颠换要高为-1 分,而颠换的得分为-5 分。
还有一种叫 BLAST 矩阵。经过大量实际比对发现,如果令被比对的两个核苷酸相同时 得分为+5 分,不相同为-4 分,这时比对效果最好。这个矩阵广泛地被 DNA 序列比较所采 用。没有为什么,就是好,实践经验所得。因为这个矩阵最早应用于 BLAST 工具,因此得 名 BLAST 矩阵。有关 BLAST 的内容将在后面的章节详细介绍。DNA 序列常见的三种替换 记分矩阵就是这三个。
二、蛋白质序列的替换记分矩阵
首先,蛋白质也有纯理论用的等价矩阵,相同得 1 分,不同得 0 分。
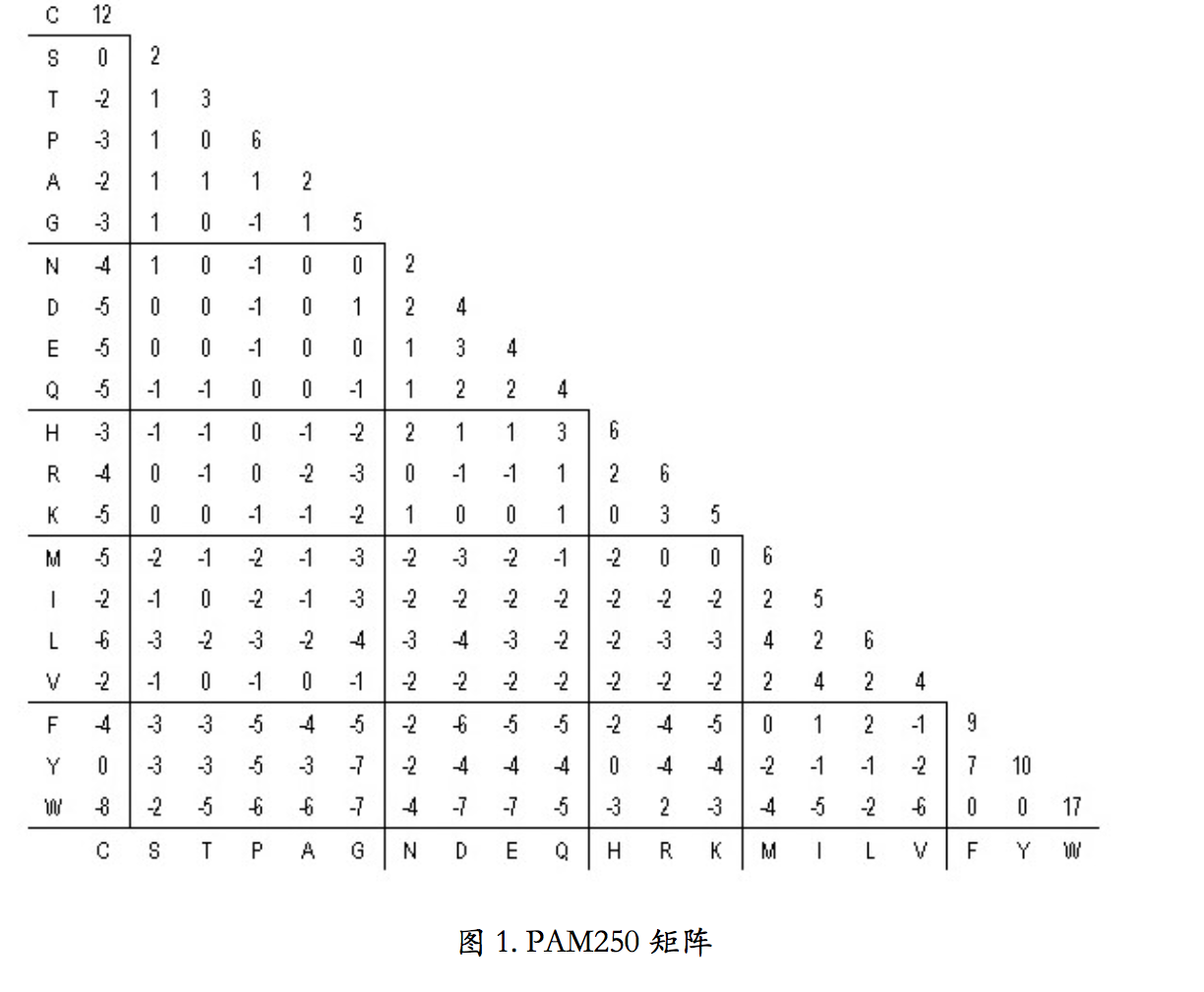
此外,蛋白质最常用的两种矩阵是 PAM 矩阵和 BLOSUM 矩阵。PAM 矩阵基于进化原 理。如果两种氨基酸替换频繁,说明自然界容易接受这种替换,那么这一对氨基酸替换的得 分就应该高。PAM 矩阵是目前蛋白质序列比较中最广泛使用的记分方法之一。基础的 PAM-1 矩阵反应的是进化产生的每一百个氨基酸平均发生一个突变的量值,由统计方法得到。 PAM-1 自乘 n 次,可以得到 PAM-n ,表示发生了更多次突变。我们需要根据要比较的序列 之间的亲缘关系远近,来选择适合的 PAM 矩阵。如果序列亲缘关系远,也就是说序列间会 有很多突变,那就选 PAM 后面跟一个大数字的矩阵。如果亲缘关系近,也就是突变比较少, 序列间大多数地方都是一样的,那就选 PAM 后面跟一个小数字的矩阵。
图 1 是 PAM250 矩阵。对角线上的数值为匹配氨基酸的得分。其他位置上≥0 的得分代 表对应的一对氨基酸为相似氨基酸,<0 的是不相似的氨基酸。
BLOSUM 矩阵有和 PAM 矩阵相同的地方,也有不同的地方。相同的是,BLOSUM 矩阵 后面也带有一个编号,有很多种 BLOSUM 矩阵。不同的是,BLOSUM 矩阵都是通过对大量 符合特定要求的序列计算而来的。这点和 PAM 矩阵不同的。PAM-1 矩阵是基于相似度大于 85%的序列计算产生的,也就是通过关系较近的序列计算出来的。那些进化距离较远的矩阵, 如 PAM-250,是通过 PAM-1 自乘得到的。也就是说,BLOSUM 矩阵的相似性是根据真实数 据产生的,而 PAM 矩阵是通过矩阵自乘外推而来的。和 PAM 矩阵的另一个不同之处是 BLOSUM 矩阵的编号。这些编号,比如 BLOSUM80 中的 80,代表这个矩阵是由一致度≥80% 的序列计算而来的。同理,BLOSUM62 是指这个矩阵是由一致度≥62%的序列计算而来的。 因此,BLOSUM 后面跟一个小数字的矩阵适合用于比较相似度低的序列,也就是亲缘关系 远的序列;而 BLOSUM 后面跟一个大数字的矩阵适合比较相似度高的序列,也就是亲缘关 系近的序列。
图 2 是 BLOSUM 62 矩阵.样子和 PAM 矩阵差不多,但是里面的数值是不一样的。同样, ≥0 的得分代表对应的一对氨基酸为相似氨基酸,<0 的是不相似的氨基酸。

现在我们总结一下到底是用 PAM 几,或者 BLOSUME 几。PAM1 对应的氨基酸差异是 1%,这是基础矩阵,由实际数据计算得出。而 PAM11 是由 PAM1 自乘 11 次得到的,他对 应的氨基酸差异可不是 11%,而是大约在 10%左右(图 3)。同样,PAM80 对应的差异也不 是 80%,而是在 50%左右。如果你要比对的序列亲缘关系远,比如氨基酸差异在 80%左右,那就得选 PAM 自乘次数非常多的矩阵,适合的是 PAM246。但是现成的 PAM 矩阵也不是什 么号的都有,只有几个关键号的。比如这个 PAM246 就没有,有的是 PAM250。再来看 BLOSUME。BLOSUME 后面的号和 PAM 刚好相反,因为它对应的是序列的相似度。差异 在 80%左右意味着相似度在 20%左右,所以这个档次上的序列适合用的 BLOSUM 矩阵就是 BLOSUM20。概括的说,PAM 后面的数体现的是序列的差异度,但不直接等于差异度,只 是成对应关系而已;BLOSUM 后面的数体现是的序列的相似度并且直接等于相似度。所以 我们看到,随着差异度的增大,适用的 PAM 矩阵后面的编号是增大的,而 BLOSUM 矩阵 后面的编号是减小的。

进一步总结一下(图 4),亲缘关系较近的序列之间的比较,用 PAM 数小的矩阵或 BLOSUM 数大的矩阵;而亲缘关系较远的序列之间的比较,用 PAM 数大的矩阵或 BLOSUM 数小的矩阵。

接下来,问题又来了,那到底是用 PAM 还是 BLOSUM 呢?对于关系较远的序列之间 的比较,由于 PAM250 是通过矩阵自乘推算而来的,所以其准确度受到一定限制。相比之下
BLOSUM 矩阵更具优势。对于关系较近的序列之间的比较,用 PAM 或 BLOSUM 矩阵做出 的比对结果,差别不大。如果关于要比较的序列你不知道亲缘关系远近,那么就闭着眼睛用 BLOSUM62 吧!至此,如果你记不住或者听不懂前面讲的种种,只记住 BLOSUM62 这个名 字,也可以走遍天下全不怕!
除了 PAM 和 BLOSUM 矩阵,还有两个蛋白质的替换记分矩阵。一个是遗传密码矩阵 (图 1),它是通过计算一个氨基酸转换成另一个氨基酸所需的密码子变化的数目而得到的。 矩阵的值对应为据此付出的代价。如果变化一个碱基就可以使一个氨基酸的密码子转换为另 一个氨基酸的密码子,则这两个氨基酸的替换代价为 1;如果需要 2 个碱基的改变,则替换 代价为 2;再比如从蛋氨酸(Met)到酪氨酸(Tyr)三个密码子都要变,则代价为 3。遗传 密码矩阵常用于进化距离的计算,它的优点是计算结果可以直接用于绘制进化树,但是它在 蛋白质序列比对,尤其是相似程度很低的蛋白质序列比对中,很少被使用。

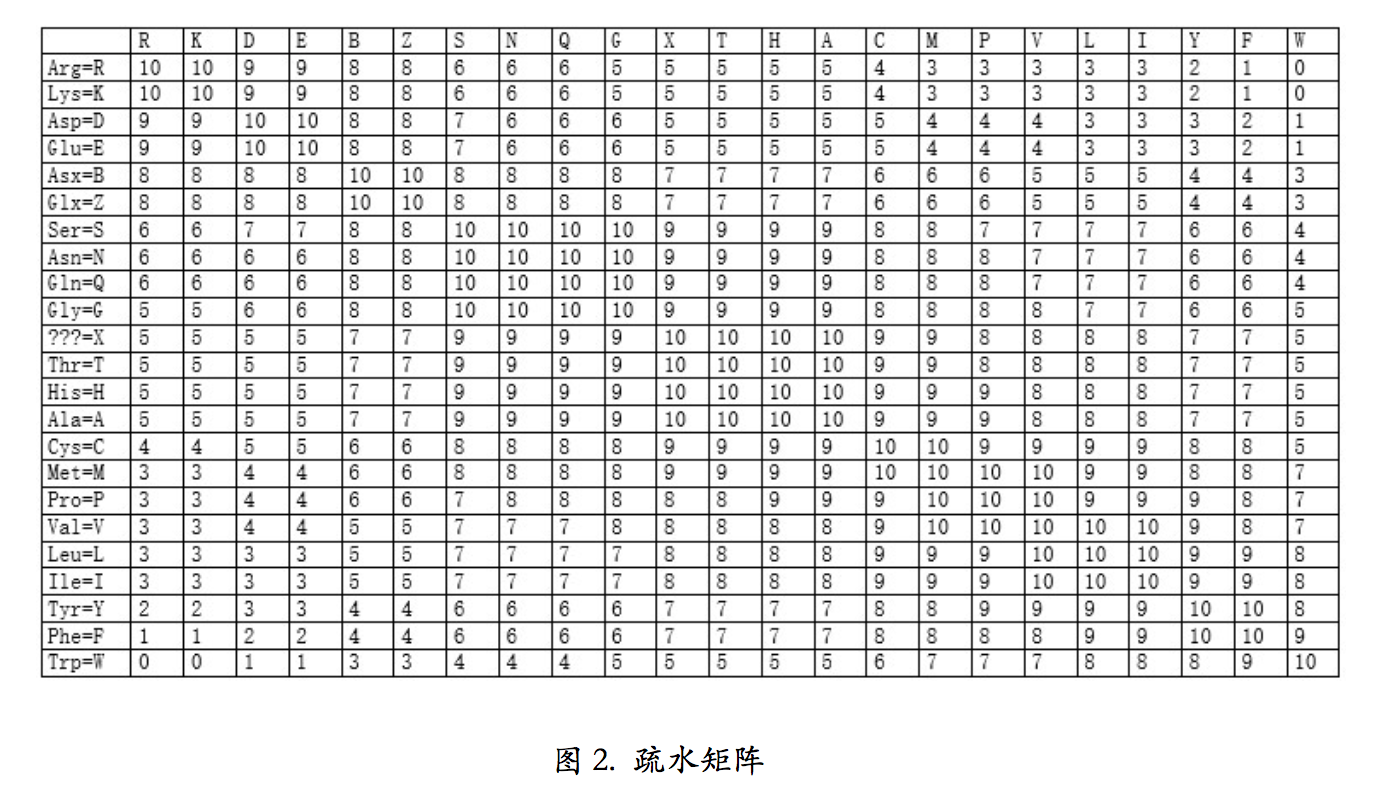
另一个疏水矩阵(图 2),它是根据氨基酸残基替换前后疏水性的变化而得到的矩阵。 若一次氨基酸替换导致疏水特性不发生太大的变化,则这种替换得分高,否则替换得分低。 疏水矩阵物理意义明确,有一定的理化性质依据,适用于偏重蛋白质功能方面的序列比对。 在这个矩阵里,氨基酸按照亲疏水性排列。前边是亲水的,后面是疏水的。
有了替换记分矩阵,就可以知道哪些氨基酸是相似的。比如下面这个例子里:

我们可以从替换记分矩阵中读出 I 和 L 相似,K 和 L 不相似。因此,它们的相似度就是 2 个相同的加上 1 个相似的,除以长度 4,等于 75%。
接下来新的问题又来了,要是两个序列的长度不相同,怎么计算它们的一致度和相似度 呢?比如下面这两条序列:

2.2 PAM矩阵与BLOSUM矩阵的区别
对于蛋白质序列,计分矩阵主要用于记录在做序列比对时两个相对应的残基的相似度,一旦这个矩阵定义好了以后,比对程式就可以利用这个矩阵,尽量将相似的残基排在一起,以达到最好的比对。
得分矩阵主要有两种,第一种就是PAM(Point Accepted Multation),另一种就是BLOSUM。
PAM矩阵(Point Accepted Mutation)
基于进化的点突变模型,如果两种氨基酸替换频繁,说明自然界接受这种替换,那么这对氨基酸替换得分就高。一个PAM就是一个进化的变异单位, 即1%的氨基酸改变,但这并不意味100次PAM后,每个氨基酸都发生变化,因为其中一些位置可能会经过多次突变,甚至可能会变回到原来的氨基酸。
PAM矩阵的制作步骤:
- 构建序列相似(大于85%)的比对
- 计算氨基酸 j 的相对突变率mj(j被其它氨基酸替换的次数)
- 针对每个氨基酸对 i 和 j , 计算 j 被 i 替换次数
- 替换次数除以相对突变率(mj)
- 利用每个氨基酸出现的频度对j 进行标准化
- 取常用对数,得到PAM-1(i, j)
- 将PAM-1自乘N次,可以得到PAM-N。
这种矩阵的缺点是一旦PAM1的矩阵有效地误 差,那么自乘250后得到的PAM250矩阵的误差就会变得很大。
如,PAM120矩阵用于比较相距120个PAM单位的序列。
一个PAM-N矩阵元素(i,j)的值: 反应两个相距N个PAM单位的序列中第i种氨基酸替换第j种氨基酸的频率。
针对不同的进化距离采用PAM 矩阵
序列相似度 = 40% 50% 60%
| | |
打分矩阵 = PAM120 PAM80 PAM60
PAM250 → 14% – 27%
BLOSUM 矩阵
此矩阵与PAM矩阵的不同之处在于:
1)用于产生矩阵的蛋白质家族及多肽链数目,BLOSUM比PAM大约多20倍。
2)PAM:家族内成员相比,然后把所有家族中对某种氨基酸的比较结果加和在一起,产生“取代”数据(PAM-1 );PAM-1自乘n次,得PAM-n。 BLOSUM:首先寻找氨基酸模式,即有意义的一段氨基酸片断(如一个结构域及其相邻的两小段氨基酸序列) ,分别比较相同的氨基酸模式之间氨基酸的保守性(某种氨基酸对另一种氨基酸的取代数据),然后,以所有 60%保守性的氨基酸模式之间的比较数据为根据,产生BLOSUM60;以所有80%保守性的氨基酸模式之间的比 较数据为根据,产生BLOSUM80。
3)PAM-n中,n 越小,表示氨基酸变异的可能性越小;相似的序列之间比较应该选用n值小的矩阵,不太相似 的序列之间比较应该选用n值大的矩阵。PAM-250用于约20%相同序列之间的比较。BLOSUM-n中,n越小,表示氨基酸相似的可能性越小;相似的序列之间比较应该选用 n 值大的矩阵,不太相似的序列之间比较应该选 用n值小的矩阵。BLOSUM-62用来比较62%相似度的序列,BLOSUM-80用来比较80%左右的序列。
讨论
BLOSUM和PAM矩阵位置
https://www.ncbi.nlm.nih.gov/IEB/ToolBox/C_DOC/lxr/source/data/
参考资料
- 山东大学 生物信息学课题组荣誉出品 http://www.crc.sdu.edu.cn/bioinfo 巩晶老师课件
- https://www.cnblogs.com/qianjinhui/archive/2013/12/29/3496628.html
